| 酒精性肝炎自噬关键基因的筛选及生物信息学分析 |
| |
| 引用本文: | 袁超, 练庆海, 尼贝贝, 等. 酒精性肝炎自噬关键基因的筛选及生物信息学分析[J]. 器官移植, 2024, 15(1): 90-101. doi: 10.3969/j.issn.1674-7445.2023163 |
| |
| 作者姓名: | 袁超 练庆海 尼贝贝 许燕 张彤 张剑 |
| |
| 作者单位: | 1.510630 广州,中山大学附属第三医院肝脏外科暨肝移植中心;2.疫苗研究所;3.生物治疗中心 |
| |
| 基金项目: | 广东省自然科学基金面上项目(2019A1515011850、2022A1515012224); |
| |
| 摘 要: | 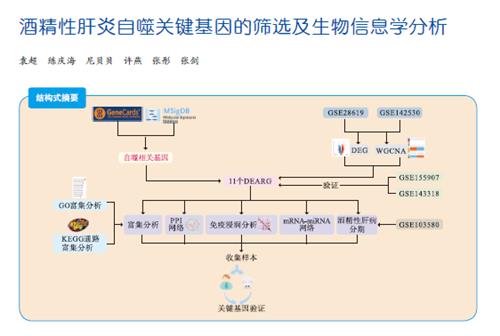
目的 筛选酒精性肝炎(AH)的自噬关键基因,探讨AH潜在的生物标志物和治疗靶点。 方法 采用基因表达综合数据库(GEO)中的2个AH基因芯片和从MSigDB、GeneCards数据库中获得的自噬相关数据集,通过加权基因共表达网络分析(WGCNA)获取关键基因。对筛选的关键基因进行基因本体(GO)、京都基因和基因组百科全书(KEGG)功能富集分析,蛋白质相互作用(PPI)分析,免疫浸润分析,构建信使RNA(mRNA)-微小RNA(miRNA)网络,进行酒精性肝病不同分期的自噬相关关键基因的表达差异分析,并进一步通过实时荧光定量逆转录聚合酶链反应(RT-qPCR)在AH患者和小鼠肝脏组织中验证。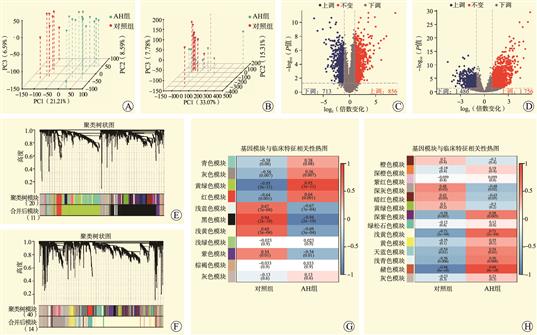
结果 本研究筛选得到了11个与AH自噬相关的基因(EEF1A2、CFTR、SOX4、TREM2、CTHRC1、HSPB8、TUBB3、PRKAA2、RNASE1、MTCL1、HGF),均为上调基因。在AH患者和小鼠肝脏组织中,SOX4、TREM2、HSPB8、PRKAA2在AH组中的相对表达量均高于对照组。 结论 SOX4、TREM2、HSPB8、PRKAA2可能是AH潜在的生物标志物和治疗靶点。

|
| 关 键 词: | 酒精性肝炎 自噬 关键基因 生物信息学 加权基因共表达网络分析(WGCNA) 基因本体(GO) 京都基因和基因组百科全书(KEGG) 蛋白质相互作用(PPI) |
| 收稿时间: | 2023-08-24 |
|
| 点击此处可从《器官移植》浏览原始摘要信息 |
|
点击此处可从《器官移植》下载免费的PDF全文 |
|



