| 基于机器学习的结直肠癌微卫星不稳定基因挖掘及其应用价值分析 |
| |
| 作者姓名: | 李秀勤 韩腾辉 王帅 沈刚 朱军 |
| |
| 作者单位: | 1.中国人民解放军南部战区空军医院 普通外科, 广东 广州 510000;2.中国人民解放军空军军医大学西京医院 神经内科,陕西 西安 710000;3.中国人民解放军空军西安飞行学院一旅明港场站医院 门诊部,河南 信阳 463200 |
| |
| 基金项目: | 国家自然科学基金资助项目(82100680)。 |
| |
| 摘 要: | 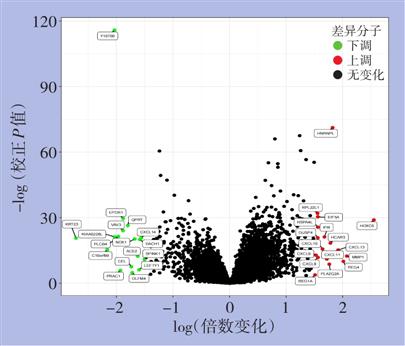
背景与目的 结直肠癌(CRC)是全球第三大最常诊断的恶性肿瘤和第二大癌症死亡原因。最新指南推荐所有的CRC患者需要进行微卫星不稳定(MSI)的检测。MSI患者往往具有错配修复蛋白缺失(dMMR)。MSI/dMMR状态已被用作生物标志物预测对免疫治疗的有利反应和预后。然而MSI特征基因及其与肿瘤浸润的免疫细胞的关系未进行阐述。因此本研究通过使用机器学习的方式发掘CRC中新型的MSI特征基因,并且验证其的诊断价值及其与免疫细胞浸润的关系。方法 按照纳入排除标准,将GEO数据库中GSE39582数据集作为训练集,将TCGA数据库中COAD数据集作为外部验证集。使用机器学习的方法(LASSO回归、SVM-RFE算法),在GSE39582结直肠癌数据集中筛选MSI特征基因,并在TCGA结直肠癌数据中进行验证。采用受试者工作特征(ROC)曲线和曲线下面积(AUC)评价基因对MSI的诊断效能。CIBERSORT算法评估肿瘤样本浸润的免疫细胞成分,Spearman相关性分析验证MSI特征基因和免疫细胞的关系。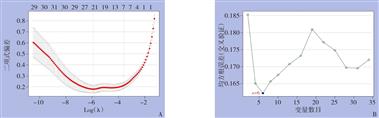
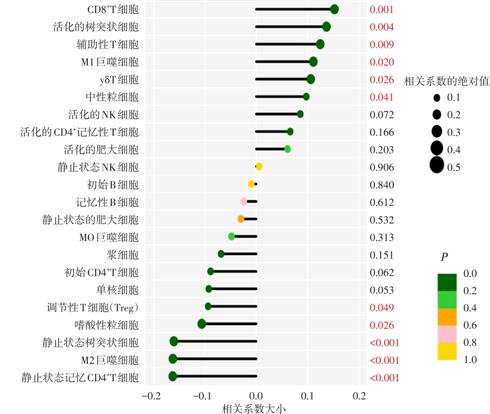
结果 训练集共纳入536例CRC患者,其中高频MSI(MSI-H)77例(14.37%)。在验证集中,共计389例CRC患者,其中MSI-H 67例(17.22%)。基线资料分析显示,MSI-H/dMMR CRC的TNM分期存活率优于低频MSI(MSI-L)或微卫星稳定(MSS)/错配蛋白完整(pMMR)CRC(P<0.05)。在GSE39582数据集中,LASSO回归筛选MSI特征基因21个,SVM-RFE算法筛选基因6个,结合两种算法确定MSI特征基因为EIF5A、CXCL13、HNRNPL、HOXC6、RPL22L1、Y16709。在TCGA数据库中进一步验证MSI特征基因的诊断效能,研究发现EIF5A的诊断效能最高。在训练集和验证集中,EIF5A的AUC值分别为0.922和0.805。同时,Spearman相关性分析发现,EIF5A主要与CD8+T细胞,活化的树突状细胞,辅助性T细胞,M1型巨噬细胞,γδT细胞,中性粒细胞成正相关;与CD4+记忆性T细胞,M2型巨噬细胞,静止树突状细胞,嗜酸性粒细胞,调节性T细胞呈负相关。结论 CRC的新型MSI特征基因分析结果表明,EIF5A对CRC MSI的诊断具有较好的诊断作用和临床价值,同时提示EIF5A与免疫细胞及免疫微环境相关。因此,EIF5A可能成为免疫检查点治疗的新型标志物。
|
| 关 键 词: | 结直肠肿瘤 微卫星不稳定性 DNA错配修复 淋巴细胞,肿瘤浸润 机器学习 |
| 收稿时间: | 2021-11-29 |
| 修稿时间: | 2022-04-18 |
|
| 点击此处可从《中国普通外科杂志》浏览原始摘要信息 |
|
点击此处可从《中国普通外科杂志》下载免费的PDF全文 |
|



